近日,《Cell Discovery》(IF:12.5)刊登了题为“scCirclehunter delineates ecDNA-containing cells using single-cell ATAC-seq, with a focus on glioblastoma”的研究论文。该论文以苏州大学基础医学院为第一单位发表,其中硕士研究生姜戎为第一作者,黄茉莉教授为最后通讯作者。该研究开发了一个名为scCirclehunter的新型计算框架,实现了在单细胞分辨率下精确鉴定染色体外DNA(ecDNA)并深入分析了其在胶质母细胞瘤(GBM)中的调控异质性 。

在癌症研究中,染色体外DNA(ecDNA)被认为是驱动肿瘤异质性和耐药性的关键因素,且与患者的预后不良密切相关。尽管ecDNA在多种癌症(如GBM的ecDNA检出率高达60%)中普遍存在,但以往的研究大多依赖Bulk测序,难以捕捉到患者体内不同细胞间ecDNA的动态分布及其复杂的转录调控差异。因此,在单细胞水平上精准识别携带ecDNA的细胞群,对于理解肿瘤演化具有重要意义 。
这项合作研究基于scATAC-seq开发了scCirclehunter算法框架,通过高斯混合模型(GMM)或k-means聚类将ecDNA准确分配到具体的细胞群体中。通过对多名GBM患者的数据分析,研究团队揭示了ecDNA在不同患者间及同一肿瘤患者不同空间区域内的显著异质性。并以ecNR2E1为例,通过整合单细胞转录组(scRNA-seq)数据,发现ecDNA能够通过构建全新的转录因子调节网络,异常激活下游致癌基因(如CD24、FOXO3等),从而推动GBM进展和免疫逃逸(图)。
scCirclehunter为研究患者特异性的ecDNA提供了一个经济且高效的单细胞精度分析工具。该研究不仅深化了我们对ecDNA驱动肿瘤异质性机制的理解,也为未来开发针对ecDNA的精准靶向疗法提供了重要理论依据 。
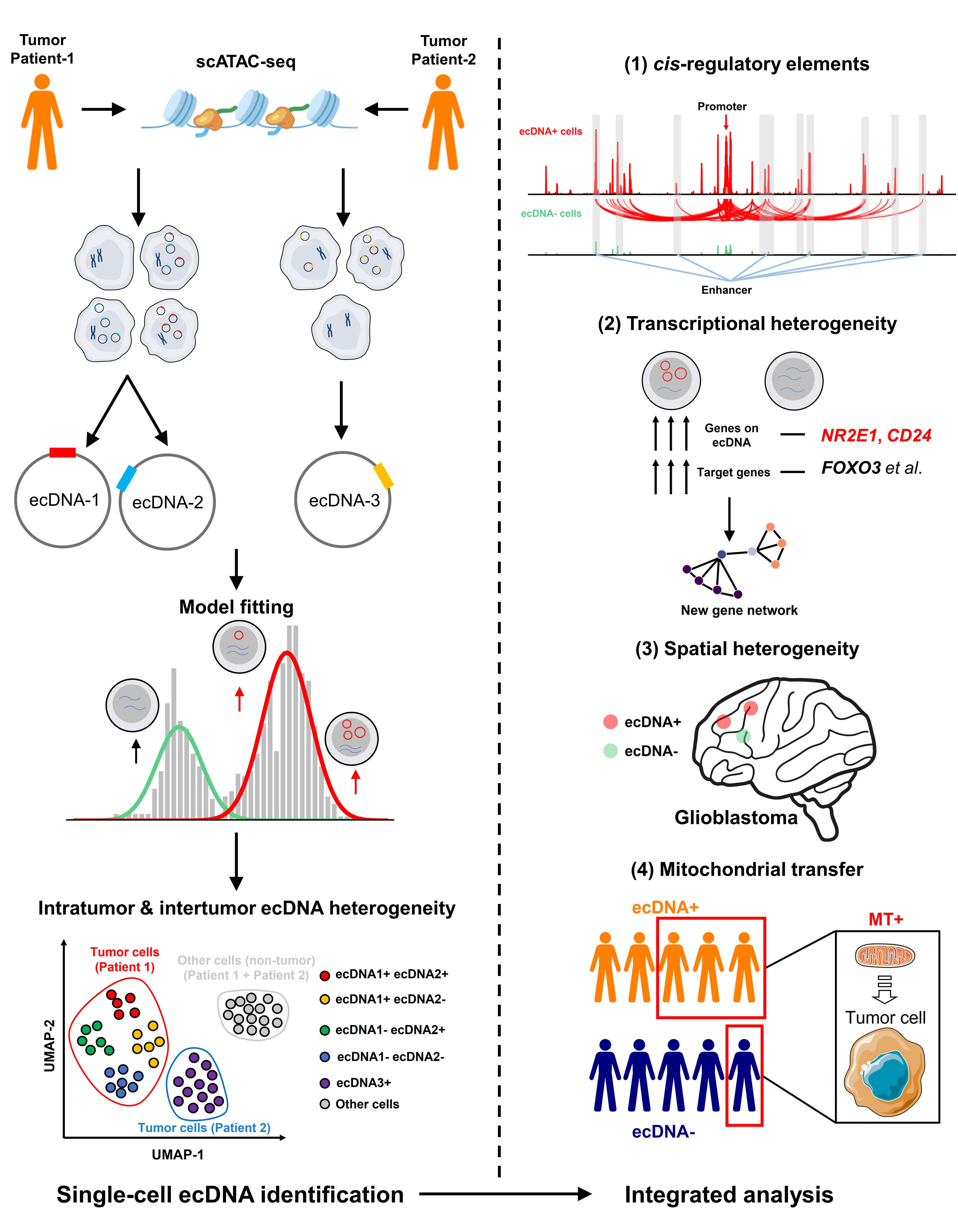
图scCirclehunter计算框架及对GBM ecDNA的分析示意图
本研究得到了国家自然科学基金面上项目和重点研发计划的资助。
原文链接:https://www.nature.com/articles/s41421-025-00842-9

